(图文|金炜炜 审核人|龚静)近日,公司龚静教授研究团队在《Nucleic Acids Research》杂志发表题为《Animal-eRNAdb: a comprehensive animal enhancer RNA database》的数据库文章。该文章对10种动物不同组织的增强子RNA(eRNA)进行了深入分析,发现了大量组织特异表达的eRNAs,并构建了在线数据库,方便其他研究者使用。龚静教授和上海复旦大学张朝教授为共同通讯作者,公司博士金炜炜和硕士蒋光辉为共同第一作者。
增强子RNA(Enhancer RNAs, eRNAs)是由增强子转录形成的一类非编码RNA。转录组学和(表观)基因组学最新研究进展表明,活性增强子可以打开局部染色质结构并暴露DNA基序以吸引转录因子 (TF),这些TF进一步招募RNA聚合酶以生成非编码 RNA (ncRNA),其被定义为增强子RNA (eRNA)。在早期研究中,eRNA仅被视为增强子转录的副产品,没有特定功能。随着对 eRNA 的日益关注,越来越多的证据表明,eRNA的表达不仅与增强子的活性高度相关,还与基因调控及疾病发生发展有着重要的关系。例如,eRNA OLMALINC可以通过调节硬脂酰辅酶A去饱和酶来影响体重。由于eRNA的重要性,已经在各种人体组织中鉴定了许多eRNA,并已开发数个人类的eRNA数据库如HeRA、eRic和TCeA。然而,eRNA在动物中的表达情况尚未完全阐明。此外,许多增强子高度保守,有些甚至在长进化距离内是保守的。因此,对保守eRNA的进一步研究有助于更好地了解不同物种中增强子和eRNA的潜在功能。总之,eRNA的表达图谱和其之间的保守性研究对揭示基因表达调控和动物表型的潜在机制具有重要价值。
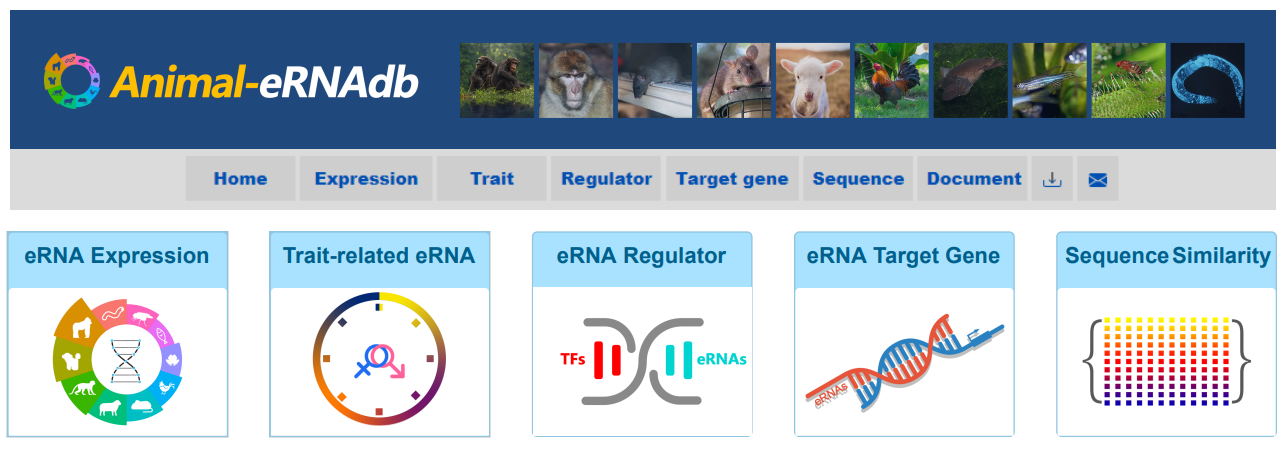
本研究中,团队成员收集了10个物种5085个样本的RNA-seq数据,通过生物信息学计算方法,一共鉴定185,177 eRNAs。同时针对这些eRNAs,团队成员也对其可能的关联性状、转录因子和目标基因做了鉴定。此外,为了探索不同物种eRNA的功能,团队成员还对不同物种间的eRNA的序列保守性进行了分析,希望对eRNA的功能解析提供更多的线索。基于上述分析结果,构建了Animal-eRNAdb数据库(http://gong_lab.hzau.edu.cn/Animal-eRNAdb/)。该数据库提供了5个模块,方便其他研究者浏览和查询eRNA相关的内容。该数据库的构建,为动物eRNA研究提供了丰富的资源,有利于动物调控机制的研究。
龚静教授从2018年到太阳成集团官网组建团队以来,一直希望能和学校的动植物等优势学科相融合。通过文献调研,她发现动物大样本、多组学相关的研究及数据库构建远远落后于对人类的研究,且多物种综合性数据库非常少。因此,龚静教授决定在“动物多组学数据分析及数据库构建”方向开展研究。通过数年努力,课题组先后收集了多个动物的几千套基因组和转录组数据,通过数据分析,先后构建了Animal-ImputeDB(Nucleic Acids Research,2019)、Animal-APAdb(Nucleic Acids Research,2020)、Animal-eRNAdb(Nucleic Acids Research,2021)等多个跨物种生物信息数据库。后期,龚静教授将继续在此方向开展研究,并希望能和相关研究学者建立合作,一起促进该方向的发展。
英文摘要:
Enhancer RNAs (eRNAs) are a class of non-coding RNAs transcribed from enhancers. As the markers of active enhancers, eRNAs play important roles in gene regulation and are associated with various complex traits and characteristics. With increasing attention to eRNAs, numerous eRNAs have been identified in different human tissues. However, the expression landscape, regulatory network and potential functions of eRNAs in animals have not been fully elucidated. Here, we systematically characterized 185 177 eRNAs from 5085 samples across 10 species by mapping the RNA sequencing data to the regions of known enhancers. To explore their potential functions based on evolutionary conservation, we investigated the sequence similarity of eRNAs among multiple species. In addition, we identified the possible associations between eRNAs and transcription factors (TFs) or nearby genes to decipher their possible regulators and target genes, as well as characterized trait-related eRNAs to explore their potential functions in biological processes. Based on these findings, we further developed Animal-eRNAdb (http://gong_lab.hzau.edu.cn/Animal-eRNAdb/), a user-friendly database for data searching, browsing and downloading. With the comprehensive characterization of eRNAs in various tissues of different species, Animal-eRNAdb may greatly facilitate the exploration of functions and mechanisms of eRNAs.
文章地址:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab832/6374163
